Резюме
Передумови
Перетин штучного інтелекту (ШІ) з дослідженнями раку зростає, і багато досягнень зосереджені на аналізі зображень раку.
Мета
Описати та узагальнити літературу щодо діагностичної точності ШІ в ранній візуалізаційній діагностиці раку шийки матки відповідно до Preferred Reporting Items for Systematic Reviews and Meta-Analyses Extension for Scoping Reviews (PRISMA-ScR).
Стратегія пошуку
Було використано методологію Arksey та O’Malley, а пошук у базах даних PubMed, Scopus та Google Scholar проводився з використанням комбінації ключових слів англійською та іспанською мовами.
Критерії відбору
Виявлені назви та резюме були переглянуті для відбору оригінальних звітів та перехресної перевірки на наявність збігів у випадках.
Збір та аналіз даних
Описовий підсумок був організований за алгоритмом ШІ, що використовувався, загальною кількістю проаналізованих зображень, джерелом даних, клінічними критеріями порівняння та результативністю діагностики.
Основні результати
Ми відібрали 32 дослідження, опубліковані між 2009 і 2022 роками. Основними джерелами зображень були цифрова кольпоскопія, цервікографія та мобільні пристрої. Алгоритми машинного навчання/глибокого навчання (ГН), застосовані в статтях, включали метод опорних векторів (МОВ), класифікатор випадкових лісів, k-найближчих сусідів, багатошаровий перцептрон, C4.5, Naïve Bayes, AdaBoost, XGboots, умовні випадкові поля, класифікатор Байєса, згорткову нейронну мережу (ЗНМ; і варіації), ResNet (кілька версій), YOLO+EfficientNetB0, і групу візуальної геометрії (ГВГ; кілька версій). Методи МОВ та ГН (ЗНМ, ResNet, ГВГ) показали найкращі результати діагностики з точністю понад 97%.
Висновок
Ми дійшли висновку, що використання ШІ для скринінгу раку шийки матки з роками зросло, а деякі результати (переважно з ГН) є дуже багатообіцяючими. Однак для підтвердження цих висновків необхідні подальші дослідження.
1 ВСТУП
Рак шийки матки є четвертою основною причиною смерті від раку серед жінок у всьому світі,1 причому 70% випадків припадає на країни, що розвиваються.2 Захворювання причинно пов’язане з персистуючою інфекцією певними онкогенними типами вірусу папіломи людини (ВПЛ), в більшості випадків з високим ризиком 16 і 18 типів.1 Адекватне впровадження програм первинної та вторинної профілактики, включаючи раннє виявлення передракових утворень і ВПЛ-інфекції за допомогою цервікально-вагінальної цитології і генотипування вірусу на основі аналізу ДНК, відповідно, могло б істотно знизити смертність.2
Цитологія має ряд обмежень. Чутливість варіюється від 52,9% до 80%, а специфічність – від 56,6% до 99,2%.3, 4 Ці відмінності в операційних характеристиках тесту можуть бути пов’язані з відмінностями між патологами і підготовкою осіб, відповідальних за забір зразків.5, 6 У деяких країнах доставка результатів пацієнтці може зайняти до 2 тижнів, що призводить до того, що великий відсоток жінок не звертаються за результатами.7 У сільських громадах з поганою інфраструктурою для цитологічного аналізу часто використовують візуальний огляд з оцтовою кислотою (VIA). Візуальний огляд за допомогою оцтової кислоти може вимагати меншого професійного досвіду, але потребує навчальних навичок для оцінки оцтовобілих ділянок, і має чутливість 53%-73% і специфічність 82%-87% для патологій шийки матки.8 Аналіз ДНК ВПЛ має високу чутливість 94% і специфічність від 84% до 90%.8 Незважаючи на продемонстровану точність первинного тестування на ВПЛ, впровадження цього методу відбувається повільно через обмежену доступність тестів, а також потребу в дуже складних лабораторіях, які дозволяють проводити ампліфікацію нуклеїнових кислот і полімеразну ланцюгову реакцію. Зокрема, його впровадження є складним у сільській місцевості та районах з низьким рівнем доходу, де спостерігаються дуже високі показники захворюваності та смертності від раку шийки матки.9–11
Діагностика шийки матки за допомогою методів, що базуються на зображеннях, незалежно від того, чи проводиться вона шляхом безпосереднього візуального огляду або за допомогою кольпоскопа, значною мірою залежить від суб’єктивного досвіду та навичок, набутих під час професійної підготовки. На сьогоднішній день золотим стандартом діагностики є кольпоскопія, а за необхідності – біопсія. Кольпоскопія сприяє збільшенню зображень, що дозволяє виявити нерівності тканин, які можуть бути непомітними для неозброєного ока. Тим не менш, на точність діагностики впливає як міжспостережна, так і внутрішньоспостережна варіабельність, а специфічність є низькою і коливається від 30% до 70%. Цифрова кольпоскопія в поєднанні з глибоким навчанням (ГН) може мати потенціал для поліпшення автоматичної класифікації зображень 12 , але все ще вимагає професійного досвіду і наявності кольпоскопів, що часто обмежено в сільській місцевості в країнах з низьким рівнем доходу. Зображення шийки матки, отримані за допомогою смартфонів і надіслані на кольпоскоп, можуть бути корисним діагностичним тестом для цих районів, але нещодавній систематичний огляд показав обмежену чутливість і специфічність.13
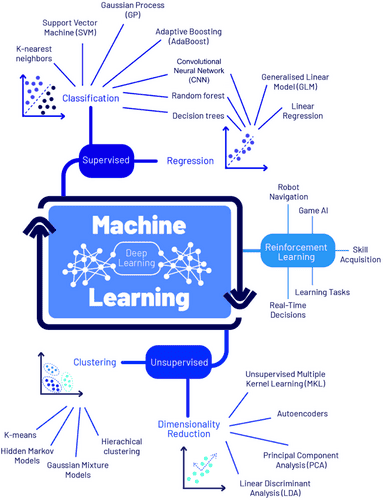
Останнім часом наукова спільнота розробляє пристрої на основі штучного інтелекту (ШІ) для гістологічної або візуальної діагностики як реальну альтернативу 13, яка може подолати вищезгадані обмеження. Діагностична візуалізація шийки матки, включаючи зону трансформації, де відбувається 87% уражень,8 може забезпечити достовірну діагностику в режимі реального часу з меншими лабораторними вимогами і без потреби у висококваліфікованому персоналі. На Зображенні 1 показані різні алгоритми ШІ, які були використані в медичних дослідженнях.
ЗОБРАЖЕННЯ 1
Класифікація алгоритмів машинного навчання.
ШІ відноситься до обчислювальних систем, які запрограмовані функціонувати подібно до людського розуму. Ці системи призначені для навчання на основі досвіду, прийняття автономних рішень і адаптації до мінливого середовища для досягнення конкретних цілей, таких як автоматична класифікація зображень 7, 8 і діагностика на основі зображень. Наприклад, машинне навчання (МН) і ГН передбачають навчання комп’ютера виконувати завдання з певною метою без явного програмування правил, як це робити.14, 15 Такий підхід може бути особливо корисним у країнах, що розвиваються, і громадах, розташованих далеко від міських центрів.
Діагностика раку шийки матки на основі застосування ШІ на зображеннях передбачає навчання керованого алгоритму (МН або ГН) на зображеннях, маркованих експертом. Під час цього навчання системи МН (інтелектуальні агенти) навчаються розрізняти зображення різних класів (нормальні або патологічні), а параметри математичних моделей, що описують ці методи, оптимізуються. Після успішного навчання алгоритми оцінюються за допомогою валідаційних зображень для вимірювання їхньої здатності до ідентифікації та узагальнення з використанням встановлених метрик класифікації, таких як точність, чутливість, специфічність, площа під кривою (робоча характеристика приймача) та оцінка F1, серед інших. Зокрема, алгоритм ШІ знаходить закономірності або дискримінаційні ознаки на зображеннях. Наприклад, згорткова нейронна мережа (ЗНМ) розбиває зображення на кілька згорткових фільтрів, здатних виявити основні та прості патерни (лінії або форми), а також більш складні патерни, такі як краї або текстури. Таким чином виділяються унікальні особливості, які використовуються для диференціації нормального зображення від патологічного. Хоча інтеграція ШІ в діагностику раку шийки матки за допомогою візуалізації зазнала значного зростання, мета полягає не в тому, щоб замінити фахівця, а в тому, щоб розробити автоматизовані, надійні і швидкі системи підтримки діагностики, які підвищують загальну ефективність.
Незважаючи на значні досягнення в галузі ШІ в медицині, застосування ШІ в діагностиці раку шийки матки ще не є широко відомим. Наразі ми зосереджені на дослідженні, спрямованому на розробку та перевірку портативної системи для скринінгу раку шийки матки (номер гранту 125189783229, 897/2021, Міністерство науки, технологій та інновацій Колумбії). Цей пристрій має намір використовувати ШІ для класифікації зображень шийки матки. Звідси випливає два дослідницьких питання: По-перше, який алгоритм ШІ було розроблено для виявлення раку шийки матки за зображеннями шийки матки? По-друге, які алгоритми є найбільш перспективними для скринінгу та діагностики раку шийки матки? Щоб відповісти на ці запитання, було проведено попередній огляд, щоб визначити та сконденсувати існуючі дані щодо точності класифікації методів МН у виявленні раку шийки матки за допомогою аналізу зображень шийки матки. Такий дизайн дослідження дозволяє вивчити широту доказової бази і визначити, чи можна проводити подальший систематичний огляд.16 Ми дотримувалися Preferred Reporting Items for Systematic Reviews and Meta-Analyses Extension for Scoping Reviews (PRISMA-ScR).
2 МАТЕРІАЛИ ТА МЕТОДИ
2.1 Тип дослідження та джерела інформації
Ми провели огляд статей, опублікованих у період з 1 січня 2009 року по 31 грудня 2022 року, за методологією Arksey and O’Malley на основі джерел PubMed, Scopus та Google Scholar. Ми використовували комбінацію іспанських та англійських ключових слів, включаючи рак шийки матки або новоутворення шийки матки, а також ШІ або МН, ГН, візуальний або прямий візуальний огляд. Крім того, ми використовували стратегію “снігової кулі” для визначення додаткових джерел з посилань на деякі повні тексти. Були використані терміни MeSH (Медичні предметні рубрики) та вільнотекстові терміни.
2.2 Критерії прийнятності
Ми включили оригінальні статті кількісних досліджень і тези конференцій, в яких чітко описано застосування МН/ГН для класифікації зображень шийки матки, отриманих за допомогою цифрової кольпоскопії, цервікографії або мобільних пристроїв, що використовуються для класифікації. Показники точності, чутливості, специфічності або AUC повинні бути ідентифіковані в кожній статті. Ми виключили статті про сегментацію зображень при раку шийки матки, оскільки вони не були присвячені застосуванню МН/ГН, статті про застосування МН/ГН в гістології та гістопатології, оглядові статті, тези, патенти, редакційні статті, листи до редактора, статті, які не мали повного тексту, а також статті, цілі дослідження яких не давали відповіді на дослідницьке питання.
2.3 Вилучення даних
Спочатку двоє дослідників визначили стратегію дослідження та оцінили, чи відповідають назви та анотації критеріям включення. Після виключення дублікатів були прочитані повні тексти для визначення потенційних критеріїв виключення. Розбіжності щодо відповідності критеріям відбору вирішувалися шляхом обговорення з іншим членом команди. Були отримані дані щодо типу алгоритму, кількості зображень, першоджерела зображень, референтного тесту, який використовувався як компаратор, та ефективності діагностики. Ми не проводили спеціальної оцінки ризику упередженості для включених досліджень. Якість доступної літератури оцінювали за математичним співвідношенням між метою та результатами дослідження.
2.4 Синтез та аналіз даних
Дані були організовані в матрицю доказів у стандартизований шаблон за допомогою Microsoft Excel 2016 (Microsoft Corporation), протестований дослідницькою групою, який відображає рік публікації, журнал, країну, використаний алгоритм МН/ГН, джерело зображень шийки матки та діагностичну ефективність. Ми використовували абсолютні цифри і відсотки для первинного опису досліджень відповідно до року публікації, мови, континенту, джерела зображень і предметної області журналу, зазначеної в Scopus або в описі веб-сторінки журналу. Ми повідомили про значення чутливості, специфічності, AUC та діагностичної точності, які є загальними показниками для оцінки ефективності тесту. Ми визначили, що алгоритм має хорошу діагностичну здатність, якщо його чутливість становить >95%, а специфічність >90%, при цьому значення ДНК ВПЛ слугують еталоном.8 Крім того, значення AUC і точність >0,90 вважалися показниками відмінної продуктивності.17, 18
3 РЕЗУЛЬТАТИ
3.1 Вибір джерел доказів
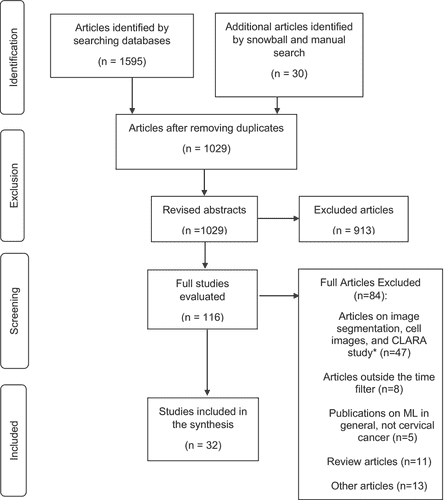
Загалом було знайдено 1625 досліджень, з яких 913 були виключені на основі анотацій, оскільки вони не відповідали меті дослідження, а 84 були виключені після прочитання повних текстів статей, оскільки вони не відповідали критеріям відбору. Нарешті, ми відібрали 32 статті, опубліковані між 2009 і 2022 роками, які описують досягнення МН/ГН для класифікації зображень шийки матки в контексті профілактики і скринінгу. На Зображенні 2 показано блок-схему включення досліджень. У Таблиці 1 наведено характеристики переглянутих статей.
ЗОБРАЖЕННЯ 2
Схема пошуку літератури зі штучного інтелекту для скринінгу раку шийки матки, 2009-2022 рр. Дослідження CLARA.19
ТАБЛИЦЯ 1. Характеристика проаналізованих досліджень: ШІ та скринінг раку шийки матки, 2009-2022 рр. (n = 32).
| Характеристики | Кількість (%) |
| Рік публікації | |
| 2009–2011 | 2 (6) |
| 2012–2014 | 2 (6) |
| 2015–2017 | 3 (9) |
| 2018–2020 | 15 (47) |
| 2021–2022 | 10 (31) |
| Джерело зображень | |
| Кольпоскопія | 20 (63) |
| Цервікограми | 5 (16) |
| Зображення шийки матки | 1 (3) |
| Кишеньковий кольпоскоп | 3 (9) |
| Смартфон | 3 (9) |
| Тематична категорія журналу | |
| Комп’ютерні науки | 13 (41) |
| Медицина | 12 (38) |
| Біомедична інженерія/інженерія/біохімія, генетика та молекулярна біологія | 5 (15) |
| Мультидисциплінарність | 2 (6) |
| Країна | |
| Китай | 7 (22) |
| Індія | 6 (19) |
| Сполучені Штати | 4 (13) |
| Південна Корея | 4 (13) |
| Японія | 3 (9) |
| Мексика | 2 (6) |
| Коста-Ріка | 2 (6) |
| Бразилія | 1 (3) |
| Венесуела | 1 (3) |
| Ефіопія | 1 (3) |
| Індонезія | 1 (3) |
- Абревіатура: ШІ, штучний інтелект.
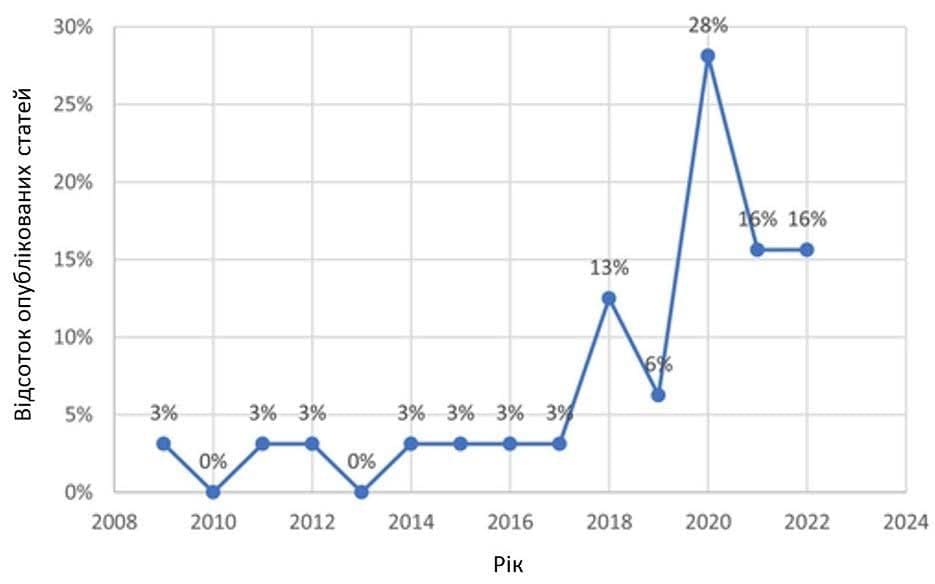
Зображення 3 ілюструє поступове зростання кількості досліджень як в МН, так і в ГН, зі значним сплеском у 2020 році для раку шийки матки та зображень шийки матки в цілому. Найбільша кількість публікацій припадає на Сполучені Штати та Індію – дев’ять і вісім відповідно. Більшість проаналізованих зображень були отримані за допомогою кольпоскопії, включаючи цифрову кольпоскопію, за нею йшли цервікограми та зображення, отримані за допомогою пристроїв на базі Android. Всі включені дослідження були опубліковані англійською мовою, більшість з них – в журналах з комп’ютерних наук (n = 13 [41%]), за якими слідували медичні журнали (n = 12 [38%]). Таким чином, вісім з 32 включених досліджень (25%) використовували ≤200 зображень для навчання та оцінки.
ЗОБРАЖЕННЯ 3
Відсоток публікацій про штучний інтелект та скринінг раку шийки матки, 2009-2022 (n = 32).
3.2 Результати ефективності методів МН та ГН у включених дослідженнях
Алгоритми класифікації, застосовані в цих дослідженнях (див. Таблицю 2), були переважно методами керованого навчання, такими як машина опорних векторів (МОВ; n = 6), випадковий ліс (n = 3), умовні випадкові поля (n = 2), C4. 5 (n = 1), Naïve Bayes (n = 3), k-найближчий сусід (KNN; n = 3), багатошаровий перцептрон (БШП; n = 1), градієнтний бустинг (n = 2), AdaBoost (n = 2), ЗНМ (та його варіації) (n = 12), ResNet (та його варіації) (n = 5), група візуальної геометрії (ГВГ; n = 2) та YOLO+EfficientNetB0 (n = 1).
ТАБЛИЦЯ 2. Опубліковані дослідження з використанням алгоритмів ШІ для класифікації раку шийки матки, 2009-2022 рр.
| Year | Journal/conference | Authors | Algorithm | Number of images | Source of images | Reference test | Diagnosis performance |
| 2022 | Scientific Reports | Kim et al.20 | ResNet-18, −50, and −101 adding segmentation information of acetowhite epithelium | 3699 | Colposcopy images | Clinical opinion or biopsy | ResNet-18
Accuracy: 74.8% Sensitivity: 65.4% Specificity: 84.3% ResNet-50 Accuracy: 76.3% Sensitivity: 69.8% Specificity: 82.9% ResNet-101 Accuracy: 74.8% Sensitivity: 72.8% Specificity: 76.9%, |
| 2022 | BMC Research Notes | Harsono et al.21 | Gradient boosting classifier | 199 patients | Smartphone-acquired VIA image (Android) | Clinical opinion | AUC = 0.85
Accuracy: 93.8% Sensitivity: 80% Specificity: 96.4% |
| 2022 | Frontiers in Pharmacology | Huang et al.22 | CNN | 10 012 | Digital colposcopy data set | Clinical opinion | Accuracy: 95.19% |
| 2022 | Medical Devices: Evidence and Research | Habtemariam et al.23 | MobileNetv2-YOLOv3 + EffecientNetB0 | 4005 + 915 | Digital colposcopy data set+histopathology images | Clinical opinion | Accuracy: 96.84% |
| 2022 | Journal of Gynecologic Oncology | Takahashi et al.24 | CNN | 167 and 593 for validation | Colposcopy images | Clinical opinion | Accuracy: 89.7% |
| 2021 | Journal of Medical Signals and Sensors | Nikookar et al.25 | Ensemble classifier with Naive Bayes, AdaBoost, random forest, random tree, SVM, decision tree | 287 | Digital colposcopy data set | Clinical opinion | Ensemble:
AUC: 0.94 Sensitivity: 96% Specificity: 94% |
| 2021 | Scientific Reports | Park et al.26 | Comparison ResNet-50 and the ML models XGB, SVM, and RF | 4119 | Cervicography images | Clinical opinion | AUCs:
ResNet-50: 0.97 XGB: 0.82 SVM: 0.84 RF: 0.79 |
| 2021 | Annals of Translational Medicine | Liu et al.27 | ResNet and combining ResNet with clinical features | 15 276 | Colposcopy images | Clinical opinion | Combined model ResNet+clinical features
AUC: 0.953 Accuracy: 88.6% Sensitivity: 93.2% Specificity: 84.6% Only ResNet AUC: 0.945 Accuracy: 88.2% Sensitivity: 90.1% Specificity: 86.7% |
| 2021 | International Journal of Medical Informatics | Yu et al.28 | GRCNN | 4753 | Colposcopy images | Clinical opinion | Accuracy: 96.87%
Sensitivity: 95.68% Specificity: 98.72% |
| 2021 | BioMed Research International | Chandran et al.29 | Two CNN: VGG-19 (TL) and CYENET | 5679 | Colposcopy photographs | Clinical opinion | VGG-19
Accuracy: 73.3% Sensitivity: 33.0% Specificity: 79.0% CYENET Accuracy: 92.3% Sensitivity: 92.4% Specificity: 96.2% |
| 2020 | Scientific Reports | Cho et al.30 | Two CNN: Inception-Resnet-v2 and Resnet-152 | 1426 | Colposcopy photographs | Clinical opinion | Resnet-v2
AUC: 0.932 Accuracy: 87.7% Sensitivity: 83.3% Specificity: 88.6% Resnet-152 AUC = 0.947 Accuracy: 87.7% Sensitivity:85.2% Specificity:88.2% |
| 2020 | IEEE J ournal of Biomedical and Health Informatics | Yue et al.31 | CNN | 4753 | Cervicograms, colposcopy | Clinical opinion | Accuracy: 96.13%
Sensitivity: 98.22% Specificity: 95.09% |
| 2020 | JMIR mHealth and uHealth | Bae et al.32 | Compare KNN, SVM, and DT | 240 | Cervicograms, smartphone-based endoscopic VIA | Clinical opinion | KNN
AUC: 0.807 Accuracy: 78.3% Sensitivity: 75.0% Specificity: 80.3% SVM AUC: 0.744 Accuracy: 74.2% Sensitivity: 72.7% Specificity: 75% DT AUC: 0.767 Accuracy: 75.8% Sensitivity: 63.6% Specificity: 82.9% |
| 2020 | Annual International Conference of the IEEE Engineering in Medicine Biology Society | Asiedu et al.33 | Evaluation of multiple combinations of contrast: VIA, VILI, GIVI | 335 | Pocket colposcope | Clinical opinion | Contrast
VIA + VILI AUC: 0.86 VIA + GIVI AUC: 0.86 VIA + VILI + GIVI AUC: 0.98 |
| 2020 | Journal of Digital Imaging | Kudva et al.34 | CNN: two pretrained networks, AlexNet and VGG-16 net and filters modified | 2198 | Cervix images | Clinical opinion | CNN-1
Accuracy: 91.46% Sensitivity: 89.16% Specificity: 93.83% CNN-2 Accuracy: 85.37% Sensitivity: 86.75% Specificity: 83.95% AlexNet Accuracy: 84.31% Sensitivity: 93.50% Specificity: 75.00% VGG-16 Accuracy: 84.15% Sensitivity: 83.13% Specificity: 85.18% |
| 2020 | BMC Medicine | Xue et al.35 | CNN
CAIADS |
101 217 | Colposcopy images and no image information | Clinical opinion | Low-grade or worse
Accuracy: 66.7% Sensitivity: 90.5% Specificity: 51.8% High-grade or worse Accuracy: 85.5% Sensitivity: 71.9% Specificity: 93.9% |
| 2020 | International Journal Cancer | Xue et al.36 | CNN: AVE for smartphone | 7587 | Smartphone-acquired VIA image | Clinical opinion | AUC >0.90 |
| 2020 | Oncology Letters | Miyagi et al.37 | CNN | 253 | Colposcopy images | Clinical opinion | AUC: 0.963
Accuracy: 94.1% Sensitivity: 95.6% Specificity: 83.3% |
| 2020 | Scientific Reports | Yuan et al.38 | Mask R-CNN + ResNet | 5384 | Colposcopy images | Clinical opinion | AUC: 0.93
Accuracy: 84.1% Sensitivity: 85.38% Specificity: 82.62% |
| 2019 | IEEE Transactions on Biomedical Engineering | Asiedu et al.39 | SVM using VIA and VILI | 200 | Pocket colposcopy | Clinical opinion | Accuracy: 80.0%
Sensitivity: 81.3% Specificity: 78.6% |
| 2019 | Journal of the National Cancer Institute | Hu et al.40 | Faster R-CNN | 2000 | Cervicography (Guanacaste study) | Clinical opinion | AUC = 0.91 |
| 2018 | Journal of Digital Imaging | Kudva et al.41 | SVM and DT | 102 | Smartphone-acquired VIA image (Android device) | Clinical opinion | SVM
Accuracy: 98.33% Sensitivity: 98.1% Specificity: 98.5% DT Accuracy: 97%–16% Sensitivity: 95% Specificity: 98.67% |
| 2018 | Proceedings of SPIE | Sapiro et al.41 | SVM | 134 | Pocket colposcope | Clinical opinion and pathology | Algorithm vs physician Accuracy: 80.6%
Sensitivity: 89.2% Specificity: 66.7% Algorithm versus pathology Accuracy: 56% Sensitivity: 75.8% Specificity: 40% |
| 2018 | International Journal of Engineering Technology Science and Research | Vasudha et al.42 | LeNet, a neural network | 690 | Digital colposcopy | Biopsy | Accuracy: 83% |
| 2018 | Oncology Letters | Sato et al.43 | Keras neural network | 485 | Digital colposcopy | Biopsy | Accuracy: 50% |
| 2017 | Computational and Mathematical Methods in Medicine | Gutierres-Fragoso et al.44 | KNN, NB, C4.5 | 200 | Digital colposcopy | Biopsy | KNN:
AUC = 0.732 NB AUC = 0.713 C4.5 AUC = 0.652 |
| 2016 | Research Journal of Pharmaceutical, Biological, and Chemical Sciences | Sukumar et al.45 | Random forest classifier | 280 | Cervicography (Guanacaste study) | Biopsy | Accuracy: 95.4% |
| 2015 | IEEE 12th International Symposium on Biomedical Imaging | Xu et al.46 | AdaBoost | 1112 | Cervicography (Guanacaste study) | Biopsy | Accuracy: 80.3% |
| 2014 | Cancer Informatics | Simoes et al.47 | Multilayer perceptron Networks | 170 | Digital colposcopy | Biopsy | AUC = 0.73
Sensitivity: 69.78% Specificity: 68% |
| 2012 | 2012 International Conference on Computing, Electronics and Electrical Technologies | Pretty Mary et al.48 | Conditional random fields | — | Digital colposcopy | Biopsy | Sensitivity: 70%
Specificity: 88% |
| 2011 | IEEE Transactions on Medical Imaging | Park et al.49 | Conditional random fields | 48a | Digital colposcopy | Biopsy | AUC = 0.8012 |
| 2009 | Computers in Biology and Medicine | Acosta-Mesa et al.50 | KNN | 38b | Digital colposcopy | Biopsy | Sensitivity: 71%
Specificity: 59% |
Скорочення: AI, штучний інтелект; AUC, площа під кривою; AVE, автоматизована візуальна оцінка; CAIADS, Кольпоскопічна допоміжна діагностична система зі штучним інтелектом; CNN, згорткова нейронна мережа; CYENET, CycleGAN та мережі перенесення стилів; DT, дерева рішень; GIVI, зображення судин із зеленим підсвічуванням; GRCNN, згорткова нейронна мережа із закритими рекурентними зв’язками; KNN, k-найближчий сусід; ML, машинне навчання; NB, Naïve Bayes; R-CNN, згорткова нейронна мережа на основі регіону; RF, випадковий ліс; SVM, машина опорних векторів; VGG, група візуальної геометрії; VIA, візуальний огляд з оцтовою кислотою; VILI, візуальний огляд шийки матки з розчином Люголя; XGB, екстремальний градієнтний бустинг.
- a Кілька зображень було отримано від 48 пацієнтів; не вказано, скільки з них було використано для навчання та валідації.
- b Десять зображень протягом перших 10 с і 300 зображень протягом наступних 5 хв для кожної жінки.
Не всі дослідження повідомили про однакові параметри діагностичної точності, і лише 11 з 32 досліджень використовували біопсію як референтний тест. Про точність повідомлялося в 22 дослідженнях, про чутливість і специфічність – в 19 дослідженнях, а про AUC – в 14 дослідженнях. Щодо чутливості, то 15,6% повідомляли про значення вище очікуваної точки відсікання (95%). Дослідження з найвищою чутливістю було засноване на МОВ, за яким слідує GRCNN, тоді як найгірша чутливість була отримана при використанні ГВГ-19, за яким слідують БШП-мережі. Що стосується специфічності, 21,9% повідомили про значення вище очікуваної точки відсікання (90%). Щодо точності або AUC, 56,2% повідомили про значення вище очікуваної точки відсікання.
4 ОБГОВОРЕННЯ
У цьому огляді ми проаналізували 32 наукові статті за 13 років (2009-2022) про використання МН, включаючи алгоритми ГН, для класифікації зображень у скринінгу раку шийки матки. Результати вказують на зростаючу тенденцію використання ГН з роками. Більшість досліджень були опубліковані в неклінічних журналах. Це підкреслює необхідність поширення цих алгоритмів у медичних журналах для сприяння клінічним дослідженням, пов’язаним з ШІ як діагностичним інструментом.
4.1 Типи алгоритмів
Для скринінгу раку шийки матки використовується декілька алгоритмів МН та ГН з контролем. Наприклад, ми зустрічаємо добре відомі МН-методи, такі як МОВ, випадковий ліс і KNN, серед інших. Крім того, ми спостерігаємо вражаючу продуктивність ГН-підходів, особливо ЗНМ та його варіацій. Нарешті, нещодавні дослідження синергетично об’єднали алгоритми МН і ГН за допомогою трансферного навчання, що підвищило чутливість і специфічність діагнозів. Ці висновки вказують на перспективні напрямки для майбутніх досліджень у цій галузі.
4.2 Продуктивність алгоритмів
Результати досліджень, проведених до цього часу, демонструють багатообіцяючі результати з точки зору діагностичної ефективності. Тим не менш, результати різняться в різних дослідженнях з точки зору специфічності, чутливості, AUC і загальної точності. З появою нових алгоритмів, дискримінаційна здатність також покращилася, особливо з 2018 року. Чутливість зросла з приблизно 70% до 2015 року до рівня, що перевищує 90% з 2018 року. Поточні дослідження з використанням методологій ГН 22–24, 26–31, 34, 37, 38 демонструють обнадійливі результати, досягаючи показників AUC, точності та чутливості, що перевищують 90%. Це відродило ентузіазм щодо застосування ГН у цій галузі. На жаль, частина досліджень представила лише показники AUC або точності для таких алгоритмів, як випадковий ліс,45 ResNet,26 і YOLO+EfficientNetB0.23 Отже, прямі порівняння стають складними через неповноту певних вимірювань. Зокрема, ЗНМ продемонстрував виняткову діагностичну ефективність у розглянутих дослідженнях, перевершуючи їх як за чутливістю, так і за специфічністю. Варто зазначити, що між чотирма опублікованими дослідженнями 31, 37, 38, 51, в яких використовувався ЗНМ, існують значні відмінності в розмірах вибірок. Помітним винятком є дослідження, проведене Hu та колегами у 2019 році,40 яке включає великий масив даних з понад 9000 цервікограм, отриманих від когорти пацієнток у Гуанакасте, Коста-Ріка. Вони застосували ЗНМ для аналізу зображень і досягли AUC 0,91. Ця AUC перевершує діагностичну точність традиційної цитології, рідинної цитології, цервікографії і навіть методів виявлення ДНК ВПЛ початкового покоління. Нарешті, можна стверджувати, що МОВ також продемонстрував багатообіцяючі результати.
Відповідно до вищесказаного, як МОВ (МН), так і ЗНМ (ГН) можуть більш ефективно виводити і розрізняти інформацію, що міститься в зображеннях шийки матки, що дозволяє автоматично і надійно ідентифікувати пацієнток з ураженнями шийки матки або навіть раком. Перевагою ЗНМ є використання фільтрів, які виділяють прості і складні ознаки із зображення в згорнутому шарі, тоді як в щільних шарах йому вдається розрізняти ці ознаки в залежності від класу. Тому можна стверджувати, що ЗНМ є одночасно і екстрактором, і класифікатором. З іншого боку, МОВ потребує окремого етапу екстракції та відбору ознак. Однак для підтвердження ефективності цієї методології в контексті скринінгових програм необхідні подальші проспективні клінічні дослідження.52 Як і очікувалося, комбінування алгоритмів призвело до покращення загальної продуктивності. Наприклад, ResNet+клінічні ознаки,27 ансамблевий класифікатор,24, 25 та Cervical Net + МОВ 21 досягли чудових показників.
4.3 Гетерогенність досліджень
Дослідження неоднорідні щодо різноманітності включених популяцій, використання різних компараторів і звітів про різні характеристики тестів. Ця неоднорідність перешкоджає прямому порівнянню алгоритмів. У деяких дослідженнях в якості компаратора використовували клінічну думку, а не золотий стандарт (біопсію)41 , що може призвести до помилки класифікації.
У деяких випадках включення пацієнток високого ризику, які вже були направлені на кольпоскопію через підозру на серйозні зміни 44, 50, не дозволяє узагальнити результати на цільову популяцію для скринінгу і може призвести до переоцінки чутливості. Крім того, кількість зображень у дослідженнях, як правило, була невеликою, за винятком дослідження Guanacaste.40 Це викликає занепокоєння, враховуючи, що рак шийки матки може мати різні макроскопічні прояви у різних пацієнтів. Дослідження, в якому було використано найбільше зображень, було розроблено Xue та співавт.35, в якому були зібрані зображення кольпоскопії та інші не кольпоскопічні зображення для навчання та оцінки.
Хоча раніше опубліковане дослідження показало, що застосування аналізу зображень для цитології шийки матки є відносно зрілим 53, наше дослідження підкреслює необхідність стандартизації дизайну та звітності про результати досліджень в області аналізу зображень шийки матки на основі МН.
У 2018 році Fernandes та співавт.54 опублікували наративний огляд, в якому надали всебічний огляд компонентів, задіяних в автоматичному аналізі зображень цифрової кольпоскопії, що може допомогти інженерам у вдосконаленні процесу отримання та аналізу зображень. Оскільки кількість публікацій про використання МН і ГН у скринінгу раку шийки матки продовжує зростати, проведення більш повної і ретельної оцінки наявних доказів може бути виправданим. Стандартизація є особливо актуальною, особливо при проведенні мета аналізу як на індивідуальному, так і на мережевому рівнях. Включення керівних принципів звітності для досліджень ШІ, таких як контрольний список Minimum Information About Clinical Artificial Intelligence Modeling (MI-CLAIM)54 , може забезпечити підвищену прозорість і точність результатів цих оглядів. Це, в свою чергу, дає змогу краще порівнювати та оцінювати різноманітні алгоритми, що використовуються. Отже, майбутні дослідження повинні прагнути до дотримання цих рекомендацій.
4.4 Обмеження огляду
Наскільки нам відомо, це перший огляд, який включає синтез діагностичних характеристик використовуваних алгоритмів, що безпосередньо пов’язано з потенційним клінічним застосуванням ШІ. Незважаючи на цей внесок, огляд має деякі обмеження. Якість доказів не була детально оцінена, оскільки автори мали на меті дослідити докази щодо використання МН і ГН для скринінгу раку шийки матки через класифікацію зображень шийки матки в загальному вигляді. Крім того, результати різних алгоритмів навчання не порівнювалися через відсутність однорідності результатів, про які повідомлялося, оскільки в деяких дослідженнях повідомлялося лише про точність, тоді як в інших – про AUC, чутливість і специфічність. Нарешті, ми не оцінювали, чи проводилася зовнішня валідація в дослідженнях, що викликає занепокоєння щодо точності діагностики, оскільки навчальні тести часто дають кращі результати.
4.5 Майбутні дослідження
Як показано в Таблиці 1, необхідно проводити спеціальні дослідження в країнах з обмеженими ресурсами і високим рівнем поширеності захворювання, включаючи такі регіони, як деякі країни Латинської Америки і Південної Африки.55 Слід зазначити, що проспективні клінічні дослідження є необхідними для оцінки реальної ефективності систем, заснованих на ГН, серед різних популяцій пацієнтів. Ці дослідження також повинні враховувати такі фактори, як економічна ефективність, доцільність і зручність використання. Крім того, етичні та регуляторні міркування повинні бути ретельно розглянуті для забезпечення відповідального використання ГН у виявленні раку шийки матки.
5 ВИСНОВОК
Результати використання алгоритмів МН/ГН для діагностичної дискримінації, особливо з точки зору чутливості, є багатообіцяючими для діагностики раку шийки матки на основі зображень шийки матки і мають потенціал бути економічно ефективними, особливо в країнах з низькими ресурсами. Тим не менш, докази залишаються непереконливими, а поточні дизайни досліджень обмежують їх застосування в клінічних сценаріях скринінгу.34 Необхідні подальші дослідження для підтвердження застосування цих технологій в клінічних умовах, включаючи оцінку популяцій пацієнтів, які беруть участь у скринінгових програмах. Серед різних алгоритмів МН/ГН найкращі результати показали ЗНМ, ResNet, YOLO+EfficientNetB0 та МОВ. Тому ми вважаємо ЗНМ та їх варіації найефективнішими методами виявлення раку шийки матки за зображеннями кольпоскопії.
ПОСИЛАННЯ НА ДЖЕРЕЛА
- Cancer (IARC) TIA for R on. Global Cancer Observatory n.d. Accessed February 22, 2023. https://gco.iarc.fr/
- Organización Panamericana de la Salud. Plan de acción sobre la prevención y el control del cáncer cervicouterino 2018–2030. 2018.
- Murillo R, Almonte M, Pereira A, et al. Cervical cancer screening pro-grams in Latin America and the Caribbean. Vaccine. 2008;26(Suppl 11):L37-L48. doi:10.1016/j.vaccine.2008.06.013
- Carrascal M, Chávez C, Sesin F. Correlación diagnóstica de la ci-tología cervical versus colposcopia en lesiones premalignas de cáncer cervicouterino. IPS Universitaria Barranquilla 2013. Biociencias. 2014;9:37-43.
- Sørbye SW, Suhrke P, Revå BW, Berland J, Maurseth RJ, Al-Shibli K. Accuracy of cervical cytology: comparison of diagnoses of 100 pap smears read by four pathologists at three hospitals in Norway. BMC Clin Pathol. 2017;17:18. d o i :10 .11 8 6 /s1 2 9 0 7- 0 17- 0 0 5 8 – 8
- Wentzensen N, Fetterman B, Castle PE, et al. p16/Ki-67 dual stain cytology for detection of cervical precancer in HPV-positive women. J Natl Cancer Inst. 2015;107:djv257. doi:10.1093/jnci/djv257
- Urrutia M-T, Poupin L. Women with cervical cancer: percep-tions about the papanicolaou test. Aquichan. 2015;15:499-507. doi:10.5294/aqui.2015.15.4.5
- Mustafa RA, Santesso N, Khatib R, et al. Systematic reviews and meta- analyses of the accuracy of HPV tests, visual inspection with acetic acid, cytology, and colposcopy. Int J Gynaecol Obstet. 2016;132:259-265. doi:10.1016/j.ijgo.2015.07.024
- Committee on Practice Bulletins—Gynecology. ACOG practice bulletin number 131: screening for cervical cancer. Obstet Gynecol. 2012;120:1222-1238. doi:10.1097/aog.0b013e318277c92a
- Yu L, Sabatino SA, White MC. Rural-urban and racial/ethnic dispari-ties in invasive cervical cancer incidence in the United States, 2010– 2014. Prev Chronic Dis. 2019;16:E70. doi:10.5888/pcd16.180447
- Li X, Deng Y, Tang W, et al. Urban-rural disparity in cancer inci-dence, mortality, and survivals in Shanghai, China, during 2002 and 2015. Front Oncol. 2018;8:579. doi:10.3389/fonc.2018.00579
- Xue P, Ng MTA, Qiao Y. The challenges of colposcopy for cervical cancer screening in LMICs and solutions by artificial intelligence. BMC Med. 2020;18:169. d o i :10 .11 8 6 /s1 2 9 1 6 – 0 2 0 – 0 1 61 3 – x
- Champin D, Ramírez-Soto MC, Vargas-Herrera J. Use of smart-phones for the detection of uterine cervical cancer: a systematic review. Cancers (Basel). 2021;13(23):6047. doi:10.3390/cancers13236047
- Garcia-Canadilla P, Sanchez-Martinez S, Crispi F, Bijnens B. Machine learning in fetal cardiology: what to expect. Fetal Diagn Ther. 2020;47:363-372. doi:10.1159/000505021
- Grant MJ, Booth A. A typology of reviews: an analysis of 14 review types and associated methodologies. Health Inf Libr J. 2009;26:91-108. doi:10.1111/j.1471-1842.2009.00848.x
- Munn Z, Peters MDJ, Stern C, Tufanaru C, McArthur A, Aromataris E. Systematic review or scoping review? Guidance for authors when choosing between a systematic or scoping review approach. BMC Med Res Methodol. 2018;18:143. d o i :10 .11 8 6 /s1 2 8 74 – 0 1 8 – 0 611- x
- Mandrekar JN. Receiver operating characteristic curve in diagnos-tic test assessment. J Thorac Oncol. 2010;5:1315-1316. doi:10.1097/JTO.0b013e3181ec173d
- Hosmer DW, Lemeshow S. Assessing the fit of the model. In: Shewhart WA, Wilks SS, Hosmer DW, Lemeshow S, eds. Applied Logistic Regression. John Wiley & Sons, Ltd; 2000:143-202. doi:10.1002/0471722146.ch5
- Hunt B, Fregnani JHTG, Brenes D, et al. Cervical lesion assessment using real-time microendoscopy image analysis in Brazil. The CLARA study. Int J Cancer. 2021;149(2):431–441. doi:10.1002/ijc.33543
- Kim J, Park C, Kim S, Cho A. Convolutional neural network-based classification of cervical intraepithelial neoplasias using colpo-scopic image segmentation for acetowhite epithelium. Sci Rep. 2022;12:17228. d o i :10 .10 3 8 /s 41 59 8 – 0 2 2- 2 1 6 9 2- 5
- Harsono AB, Susiarno H, Suardi D, et al. Cervical pre-cancerous lesion detection: development of smartphone-based VIA appli-cation using artificial intelligence. BMC Res Notes. 2022;15:356. d o i :10 .11 8 6 /s1 3 10 4 – 0 2 2- 0 6 2 5 0 – 6
- Huang W, Sun S, Yu Z, Lu S, Feng H. Chronic cervicitis and cer-vical cancer detection based on deep learning of colposcopy images toward translational pharmacology. Front Pharmacol. 2022;13:911962. doi:10.3389/fphar.2022.911962
- Habtemariam LW, Zewde ET, Simegn GL. Cervix type and cervical cancer classification system using deep learning techniques. Med Devices (Auckl). 2022;15:163-176. doi:10.2147/MDER.S366303
- Takahashi T, Matsuoka H, Sakurai R, et al. Development of a prog-nostic prediction support system for cervical intraepithelial neo-plasia using artificial intelligence-based diagnosis. J Gynecol Oncol. 2022;33(5):e57. doi:10.3802/jgo.2022.33.e57
- Nikookar E, Naderi E, Rahnavard A. Cervical cancer prediction by merging features of different colposcopic images and using ensem-ble classifier. J Med Signals Sens. 2021;11:67-78. doi:10.4103/jmss.JMSS_16_20
- Park YR, Kim YJ, Ju W, Nam K, Kim S, Kim KG. Comparison of machine and deep learning for the classification of cervical can-cer based on cervicography images. Sci Rep. 2021;11:16143. d o i :10 .10 3 8 /s 41 59 8 – 0 2 1- 9 5 74 8 – 3
- Liu L, Wang Y, Liu X, et al. Computer-aided diagnostic system based on deep learning for classifying colposcopy images. Ann Transl Med. 2021;9:1045. d o i :10 . 2 10 3 7/a t m – 2 1- 8 8 5
- Yu Y, Ma J, Zhao W, Li Z, Ding S. MSCI: a multistate dataset for col-poscopy image classification of cervical cancer screening. Int J Med Inform. 2021;146:104352. doi:10.1016/j.ijmedinf.2020.104352
- Chandran V, Sumithra MG, Karthick A, et al. Diagnosis of cervical cancer based on ensemble deep learning network using colposcopy images. Biomed Res Int. 2021;2021:e5584004. doi:10.1155/2021/5584004
- Cho B-J, Choi YJ, Lee M-J, et al. Classification of cervical neo-plasms on colposcopic photography using deep learning. Sci Rep. 2020;10:13652. d o i :10 .10 3 8 /s 41 59 8 – 0 2 0 – 7 0 4 9 0 – 4
- Yue Z, Ding S, Zhao W, et al. Automatic CIN grades prediction of sequential cervigram image using LSTM with multistate CNN fea-tures. IEEE J Biomed Health Inform. 2020;24:844-854. doi:10.1109/JBHI.2019.2922682
- Bae JK, Roh H-J, You JS, et al. Quantitative screening of cervical cancers for low-resource settings: pilot study of smartphone-based endoscopic visual inspection after acetic acid using ma-chine learning techniques. JMIR Mhealth Uhealth. 2020;8:e16467. doi:10.2196/16467
- Asiedu MN, Skerrett E, Sapiro G, Ramanujam N. Combining multi-ple contrasts for improving machine learning-based classification of cervical cancers with a low-cost point-of- care pocket colpo-scope. Annu Int Conf IEEE Eng Med Biol Soc. 2020;2020:1148-1151. doi:10.1109/EMBC44109.2020.9175858
- Kudva V, Prasad K, Guruvare S. Hybrid transfer learning for classifi-cation of uterine cervix images for cervical cancer screening. J Digit Imaging. 2020;33(3):619-631. d o i :10 .10 0 7/s10 2 7 8 – 0 19 – 0 0 2 6 9 – 1
- Xue P, Tang C, Li Q, et al. Development and validation of an artificial intelligence system for grading colposcopic impressions and guiding biopsies. BMC Med. 2020;18:406. doi:10.1186/s12916- 020- 01860- y
- Xue Z, Novetsky AP, Einstein MH, et al. A demonstration of auto-mated visual evaluation of cervical images taken with a smartphone camera. Int J Cancer. 2020;147:2416-2423. doi:10.1002/ijc.33029
- Miyagi Y, Takehara K, Nagayasu Y, Miyake T. Application of deep learning to the classification of uterine cervical squamous epithelial lesion from colposcopy images combined with HPV types. Oncol Lett. 2020;19:1602-1610. doi:10.3892/ol.2019.11214
- Yuan C, Yao Y, Cheng B, et al. The application of deep learning based diagnostic system to cervical squamous intraepithelial le-sions recognition in colposcopy images. Sci Rep. 2020;10:11639. doi:10.1038/s41598-020- 68252-3
- Asiedu MN, Simhal A, Chaudhary U, et al. Development of algo-rithms for automated detection of cervical pre-cancers with a low- cost, point- of- care, pocket colposcope. IEEE Trans Biomed Eng. 2019;66:2306-2318. doi:10.1109/TBME.2018.2887208
- Hu L, Bell D, Antani S, et al. An observational study of deep learning and automated evaluation of cervical images for cancer screening. J Natl Cancer Inst. 2019;111:923-932. doi:10.1093/jnci/djy225
- Kudva V, Prasad K, Guruvare S. Andriod device-based cervi-cal cancer screening for resource-poor settings. J Digit Imaging. 2018;31(5):646-654. d o i :10 .10 0 7/s10 2 7 8 – 0 1 8 – 0 0 8 3 – x
- Vasudha A, Mittal MJ. Cervix cancer classification using colpos-copy images by deep learning method. Int J Eng Technol Sci Res. 2018;5:426-432.
- Sato M, Horie K, Hara A, et al. Application of deep learning to the classification of images from colposcopy. Oncol Lett. 2018;15:3518-3523. doi:10.3892/ol.2018.7762
- Gutiérrez-Fragoso K, Acosta-Mesa HG, Cruz-Ramírez N, Hernández-Jiménez R. Optimization of classification strate-gies of Acetowhite temporal patterns towards improving diag-nostic performance of colposcopy. Comput Math Methods Med. 2017;2017:5989105. doi:10.1155/2017/5989105
- Sukumar P, Gnanamurthy R. Computer aided screening of cervical cancer using random forest classifier. Res J Pharm, Biol Chem Sci. 2016;7:1521-1529.
- Xu T, Kim E, Huang X. Adjustable adaboost classifier and pyra-mid features for image-based cervical cancer diagnosis. 2015 IEEE 12th International Symposium on Biomedical Imaging (ISBI). IEEE; 2015:281-285. doi:10.1109/ISBI.2015.7163868
- Simões PW, Izumi NB, Casagrande RS, et al. Classification of images acquired with colposcopy using artificial neural networks. Cancer Informat. 2014;13:119-124. doi:10.4137/CIN.S17948
- Pretty Mary D, Anandan V, Srinivasagan KG. An effective diag-nosis of cervical cancer neoplasia by extracting the diagnostic features using CRF. 2012 International Conference on Computing, Electronics and Electrical Technologies (ICCEET). IEEE; 2012:563-570. doi:10.1109/ICCEET.2012.6203885
- Park SY, Sargent D, Lieberman R, Gustafsson U. Domain-specific image analysis for cervical neoplasia detection based on condi-tional random fields. IEEE Trans Med Imaging. 2011;30:867-878. doi:10.1109/TMI.2011.2106796
- Acosta-Mesa H-G, Cruz-Ramírez N, Hernández-Jiménez R. Aceto- white temporal pattern classification using k-NN to identify pre-cancerous cervical lesion in colposcopic images. Comput Biol Med. 2009;39:778-784. doi:10.1016/j.compbiomed.2009.06.006
- Devi MA, Ravi S, Vaishnavi J, Punitha S. Classification of cervi-cal cancer using artificial neural networks. Procedia Comput Sci. 2016;89:465-472. doi:10.1016/j.procs.2016.06.105
- Rubin R. Artificial intelligence for cervical precancer screening. JAMA. 2019;321:734. doi:10.1001/jama.2019.0888
- Hou X, Shen G, Zhou L, Li Y, Wang T, Ma X. Artificial intelli-gence in cervical cancer screening and diagnosis. Front Oncol. 2022;12:851367. doi:10.3389/fonc.2022.851367
- Fernandes K, Cardoso JS, Fernandes J. Automated methods for the decision support of cervical cancer screening using digital colposcopies. IEEE Access. 2018;6:33910-33927. doi:10.1109/ACCESS.2018.2839338
- Miller KD, Siegel RL, Lin CC, et al. Cancer treatment and sur-vivorship statistics, 2016. CA Cancer J Clin. 2016;66:271-289. doi:10.3322/caac.21349